如何選擇核小體(nucleosomes)?
作者:Ellen N. Weinzapfel 博士���,Lu Sun 博士

?
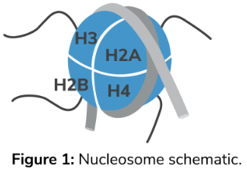
染色質(zhì)及其調(diào)控在包括癌癥等多種人類疾病中都發(fā)揮重要作用����,并共同構(gòu)成了學(xué)術(shù)���、工業(yè)和臨床研究中快速發(fā)展的研究領(lǐng)域1����。染色質(zhì)結(jié)構(gòu)的基本亞單位是核小體��,由大約147bp的DNA纏繞在核心組蛋白八聚體上構(gòu)成(圖1)2����。組蛋白亞基有多種化學(xué)修飾,如甲基化�����、乙?����;土姿峄@些修飾被稱為組蛋白修飾或翻譯后修飾(PTMs)�。
?
組蛋白修飾的功能:一種組蛋白編碼
這些修飾調(diào)節(jié)染色質(zhì)對各種蛋白質(zhì)的可及性,如轉(zhuǎn)錄因子和核小體重塑復(fù)合物3,4�。此外,組蛋白PTMs可以直接與染色質(zhì)相互作用或招募染色質(zhì)相互作用蛋白�����。因此�����,不同的組蛋白修飾與獨(dú)特的基因組功能相關(guān)�。例如,組蛋白H3賴氨酸27(H3K27me3)上的三甲基化與基因抑制密切相關(guān)5�����,而H3K4me3定位于活性轉(zhuǎn)錄起始位點(diǎn)����,因此與基因激活相關(guān)6���。
?
總之��,這些組蛋白修飾網(wǎng)絡(luò)及其動態(tài)調(diào)控協(xié)調(diào)了硬編碼基因組的表觀遺傳控制���,從而影響各種細(xì)胞過程�����,如基因調(diào)控���、細(xì)胞分化和發(fā)育4,7。
?
?
研究組蛋白修飾的理想底物
越來越多的證據(jù)表明�,核小體是體外表征許多染色質(zhì)調(diào)節(jié)因子的最佳底物。這與之前的研究不同��,之前的研究通常使用修飾的組蛋白肽來識別染色質(zhì)抑制劑或定義新的組蛋白PTM相互作用����。
?
在之前一篇關(guān)于組蛋白肽和核小體用于組蛋白PTM抗體驗證的博客文章(https://www.epicypher.com/resources/blog/histone-peptides-vs-nucleosomes-which-is-better-for-chip-antibody-validation/)中,我們已經(jīng)討論了這個主題�����。然而���,這些問題也與染色質(zhì)修飾酶的研究有關(guān)���。例如���,NSD2甲基轉(zhuǎn)移酶(與多發(fā)性骨髓瘤中的致癌重編程相關(guān))需要核小體底物才能發(fā)揮體外活性8。同樣�,當(dāng)使用重組核小體時,通過SETD8甲基轉(zhuǎn)移酶的Km顯示SAM輔因子比使用組蛋白肽底物時降低了10000倍9��。
?
隨著攜帶完全確定組蛋白修飾的重組核小體的出現(xiàn)��,為下一代染色質(zhì)研究提供了新的或更好的方法(如染色質(zhì)結(jié)合蛋白分析�、酶分析、抗體譜分析)��。
?
如何獲得重組核小體?
獲得重組核小體的制造工藝有很多種�����,如EpiCypher通過設(shè)計來獲得核小體(EpiCypher designer nucleosomes, dNucs)�����。一般來說���,大部分相關(guān)的方法基本遵循相同的一系列步驟:
?

然而�����,并非所有的重組核小體都具有相同的質(zhì)量�!不同供應(yīng)商的合成過程和質(zhì)量驗證標(biāo)準(zhǔn)可能存在很大差異�。
?
如何挑選優(yōu)質(zhì)的核小體?
如果您要使用重組核小體進(jìn)行染色質(zhì)實(shí)驗���,則需考慮以下幾點(diǎn):
?
1����、用于開發(fā)修飾組蛋白的方法�。確保使用無疤痕方法整合所需的PTMs是很重要的,這種方法可以重現(xiàn)天然組蛋白結(jié)構(gòu)���。EpiCypher 使用幾種不同的方法獲得修飾的組蛋白�,所有方法都會無疤痕整合組蛋白修飾���。
為什么這很重要呢�?許多市售的重組核小體是使用組蛋白PTM類似物構(gòu)建的����,如甲基賴氨酸類似物(MLAs) 10����,其會導(dǎo)致修飾位點(diǎn)的氨基酸序列發(fā)生變化�����。這些非天然組蛋白修飾類似物已被證明會破壞與染色質(zhì)調(diào)節(jié)蛋白和組蛋白PTM特異性抗體的相互作用�,并且這對研究生理機(jī)制來講是不理想的。因此�����,使用這些方法合成的核小體時應(yīng)格外謹(jǐn)慎11-13����。
?
2、修飾組蛋白的純度�。組裝完全定義的同質(zhì)核小體的下一步是對修飾組蛋白進(jìn)行嚴(yán)格的質(zhì)量控制。
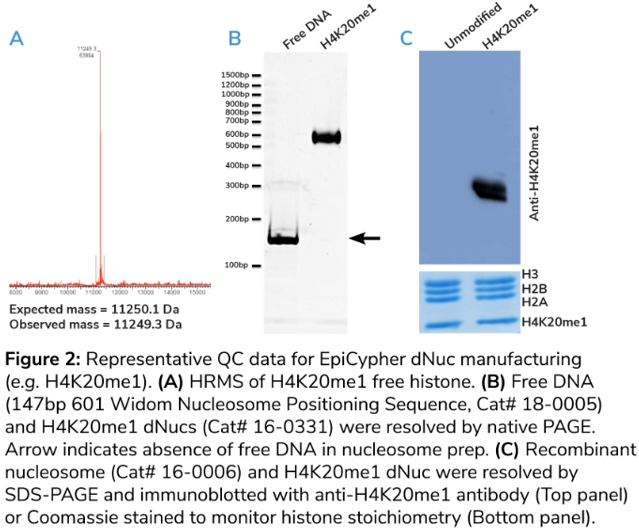
在質(zhì)控結(jié)果中�����,HPLC 跡線應(yīng)顯示是單一洗脫物質(zhì)���,表明組蛋白純度>95%���;平行高分辨率質(zhì)譜(HRMS)應(yīng)在預(yù)期質(zhì)量的1道爾頓范圍內(nèi)顯示一個單峰,沒有任何意義的額外電荷質(zhì)量(m/z)信號(例如圖2A)���。EpiCypher的所有修飾組蛋白均通過HPLC和HRMS分析進(jìn)行驗證�。
?
為什么這很重要呢��?不必要的物質(zhì)(如甲硫氨酸氧化)可能引起結(jié)構(gòu)改變�,并影響靜電相互作用或疏水相互作用,從而損害下游核小體組裝的效率����。
?
?
3、組裝核小體的質(zhì)量控制(QC)指標(biāo)�����。DNA組裝后核小體的質(zhì)量驗證對最終產(chǎn)品的信任保證至關(guān)重要��。
EpiCypher使用天然PAGE分析DNA上的dNuc組裝�����,其中使用約150bp DNA的高效組裝應(yīng)該只產(chǎn)生單一物質(zhì)����,這相對于未組裝的游離DNA���,其遷移率降低(圖2B,下圖)��。
為什么這很重要呢�?因為被污染的游離DNA會誘導(dǎo)染色質(zhì)修飾酶(如NSD2)的異常活性��,所以必須避免���。此外�����,次優(yōu)組裝可能導(dǎo)致樣品的異質(zhì)性混合�����,包括錯誤定位的核小體����。
我們還使用考馬斯染色對最終的dNucs進(jìn)行SDS-PAGE分析,以確保四種組蛋白的化學(xué)計量相等(圖2C��,下圖)�。然后��,我們通過免疫印跡證實(shí)了整合組蛋白PTM的存在(圖2C���,上圖)���。
為什么這很重要呢?如果其他種類的蛋白質(zhì)污染或偏離1:1:1:1的比例�,則可能表明組蛋白降解或組裝不良,因此有必要重新解析每個組蛋白���。而免疫印跡對于確定修飾是否存在于組蛋白的正確位置上非常重要���。
?
EpiCypher擁有令人印象深刻的產(chǎn)品目錄,其中包括83個獨(dú)特的重組核小體庫存���,并且有能力生產(chǎn)定制設(shè)計核小體���,質(zhì)量高、周期短,助力您的研究�����!
想了解更多關(guān)于EpiCypher重組核小體技術(shù)和產(chǎn)品的信息嗎��?請聯(lián)系欣博盛��!
EpiCypher核小體相關(guān)產(chǎn)品系列?
◆?Recombinant Nucleosomes
◆?SNAP-ChIP??Spike-ins
◆?SNAP-CUTANA? Spike-in Controls
◆?Modified Designer Nucleosomes (dNucs?)
◆?dCypher? Nucleosome Panels
◆?EpiDyne??Chromatin Remodeling Substrates
◆?Recombinant Nucleosomes (rNucs)
◆?Mutant Nucleosomes
◆?Purified Nucleosomes (HeLa, Avian)
◆?Nucleosome Components and Subunits
◆?Variant Nucleosomes (vNucs)
◆?Methyl DNA Designer Nucleosomes
?
參考文獻(xiàn)
1. Valencia AM, Kadoch C.?Chromatin regulatory mechanisms and therapeutic opportunities in cancer.?Nat Cell Biol, 2019. p.?(PubMed PMID: 30602726)
2. Luger K, et al.?Crystal structure of the nucleosome core particle at 2.8 A resolution.?Nature, 1997.?389(6648): p. 251-60.?(PubMed PMID: 9305837)
3. Jenuwein T, Allis CD.?Translating the histone code.?Science, 2001.?293(5532): p. 1074-80.?(PubMed PMID: 11498575)
4. Rothbart SB, Strahl BD.?Interpreting the language of histone and DNA modifications.?Biochim Biophys Acta, 2014.?1839(8): p. 627-43.?(PubMed PMID: 24631868)?(PMC4099259)
5. Cao R, et al.?Role of histone H3 lysine 27 methylation in Polycomb-group silencing.?Science, 2002.?298(5595): p. 1039-43.?(PubMed PMID: 12351676)
6. Liang G, et al.?Distinct localization of histone H3 acetylation and H3-K4 methylation to the transcription start sites in the human genome.?Proc Natl Acad Sci U S A, 2004.?101(19): p. 7357-62.?(PubMed PMID: 15123803)?(PMC409923)
7. Bernstein BE, et al.?A bivalent chromatin structure marks key developmental genes in embryonic stem cells.?Cell, 2006.?125(2): p. 315-26.?(PubMed PMID: 16630819)
8. Li Y, et al.?The target of the NSD family of histone lysine methyltransferases depends on the nature of the substrate.?J Biol Chem, 2009.?284(49): p. 34283-95.(PubMed PMID: 19808676)(PMC2797197)
9. Strelow JM, et al.?The Use of Nucleosome Substrates Improves Binding of SAM Analogs to SETD8.?J Biomol Screen, 2016.?21(8): p. 786-94.(PubMed PMID: 27369108)
10. Simon MD, et al.?The site-specific installation of methyl-lysine analogs into recombinant histones.?Cell, 2007.?128(5): p. 1003-12.?(PubMed PMID: 17350582)(PMC2932701)
11. Gloss LM, Kirsch JF.?Decreasing the basicity of the active site base, Lys-258, of Escherichia coli aspartate aminotransferase by replacement with gamma-thialysine.?Biochemistry, 1995.?34(12): p. 3990-8.?(PubMed PMID: 7696264)
12. Gellman SH.?On the role of methionine residues in the sequence-independent recognition of nonpolar protein surfaces.?Biochemistry, 1991.?30(27): p. 6633-6.?(PubMed PMID: 2065050)
13. Seeliger D, et al.?Quantitative assessment of protein interaction with methyl-lysine analogues by hybrid computational and experimental approaches.?ACS Chem Biol, 2012.?7(1): p. 150-4.?(PubMed PMID: 21991995)?(PMC3265130)
?
?
EpiCypher的注冊商標(biāo)和知識產(chǎn)權(quán)可見鏈接:https://www.epicypher.com/intellectual-property/���。
本文中的所有其他商標(biāo)和商品均為其各自公司所有�。
本文翻譯自鏈接:https://www.epicypher.com/resources/blog/finding-the-best-substrate-for-studying-histone-modifications/�����,如與原文有出入的地方��,請以英文原文為準(zhǔn)�。
未經(jīng)EpiCypher公司事先書面同意,本文件不得部分或全部復(fù)制�����。
?
關(guān)于EpiCypher公司:
EpiCypher是一家成立于2012年的表觀遺傳學(xué)公司���。從專有組蛋白肽陣列平臺EpiGold?開始�,EpiCypher開發(fā)了一系列同類產(chǎn)品。同時���,EpiCypher是重組核小體制造和開發(fā)的全球領(lǐng)導(dǎo)者�。利用其獨(dú)有技術(shù)��,不斷增加產(chǎn)品庫中高純度修飾重組核小體(dNucs?)產(chǎn)品���。dNuc?多樣性的產(chǎn)品為破譯組蛋白編碼和加速藥物開發(fā)提供了強(qiáng)大的工具。
EpiCypher還將dNuc?技術(shù)廣泛的應(yīng)用于多種分析測定產(chǎn)品中�����,包括:SNAP-ChIP?Spike-in Controls(用于抗體分析和ChIP定量)����, EpiDyne?底物(用于染色質(zhì)重塑和抑制劑篩選及開發(fā)),dCyher?測定(用于探究表觀遺傳蛋白質(zhì)-組蛋白PTM結(jié)合相互作用)�����。最近�����,EpiCypher還推出了針對ChIC、CUT&RUN和CUT&Tag的高靈敏度表觀基因組圖譜CUTANA?分析���。
?

如需了解更多詳細(xì)信息或相關(guān)產(chǎn)品����,請聯(lián)系EpiCypher中國授權(quán)代理商-欣博盛生物?
全國服務(wù)熱線: 4006-800-892? ? ? ?郵箱: market@neobioscience.com? ?
深圳: 0755-26755892? ? ? ?北京: 010-88594029? ? ? ? ?
上海: 021-34613729? ? ? ? ?廣州: 020-87615159? ? ? ? ??
代理品牌網(wǎng)站: m.yuebanme.com? ?
自主品牌網(wǎng)站: www.neobioscience.net